Por Tania Pozzo Ph.D.

Tania es una científica boliviana con un doctorado en biotecnología de la Universidad de Lund en Suecia. Ella cree que la biotecnología tiene el poder de resolver muchos problemas en nuestro mundo y permitirá a los humanos vivir en armonía con el planeta. Su objetivo es transferir conocimiento científico de nuevas tecnologías a su país de origen, razón por la cual funda iGEM Bolivia el 2019, una organización sin fines de lucro enfocada en formar jóvenes líderes en el área de biología sintética. En 2014, recibió el Premio Internacional a la Excelencia de la fundación Women in Science, establecida por UNESCO-L’Oreal. Actualmente trabaja como científica de proteínas para una compañía bio-farmacéutica en Silicon Valley, California USA.
Las personas muy pocas veces reflexionamos acerca de lo maravilloso que es el origen de nuestra naturaleza humana, si lo hiciéramos más seguido nos daríamos cuenta de que somos el producto de una evolución de aproximadamente 6 millones de años.
Nuestro cuerpo es un diseño de la naturaleza altamente complejo y muy sofisticado, estamos formados por alrededor de 30 trillones de células que coordinan en armonía todos los procesos bioquímicos necesarios para mantenernos vivos. Estas células están especializadas para brindarnos la posibilidad de actuar como biosensores con un exquisito nivel de detección, adquiriendo así «super poderes» como ser: audición, visión, olfato y el gusto. Como un ejemplo podemos mencionar que en la lengua poseemos hasta 8 mil células que tienen receptores para detectar diferentes sabores, como ser: salado, agrio, amargo, dulce y un sabor recientemente descubierto llamado umami.
La interacción entre las moléculas que dan sabor y los receptores que poseen las células en la lengua son muy específicas debido a un diseño ingenieril perfecto de la naturaleza, que al entrar en contacto mandan señales bioquímicas muy rápidas a nuestro cerebro que es el encargado de racionalizar el sabor de los alimentos. Lo aún más asombroso es que dentro de cada célula los seres humanos guardamos la información genética para que cada una de ellas lleve a cabo su tarea particular; dicha información se encuentra guardada en forma de ácido nucleico, una molécula biológica que está muy bien empacada en el núcleo de la célula, y que si la desempacamos tendría como 2 metros de largo.
En la Figura 1 se describe paso a paso cómo la información genética es transcrita y traducida desde ácido nucleico a proteína, un mecanismo que normalmente ocurre dentro de la célula, proceso conocido como el dogma central de la biología.

El dogma central de la biología tiene dos partes fundamentales: la transcripción que se encuentra indicada con flechas sólidas y la parte de la traducción que está indicada en flechas punteadas.
La célula animal es clasificada como eucariota ya que posee un núcleo donde guarda de forma compacta al ácido nucleico que enrolla a unas proteínas llamadas histonas. El ácido nucleico tiene la forma de una doble hélice unida por unos puentes de nucleótidos, donde la adenina (A) se une a la timina (T) y la guanina (G) a la citosina (C). Esta doble hélice se traduce a una sola cadena de RNA donde se reemplaza la T por un uracilo (U).
La síntesis de la proteína ocurre cuando 3 nucleótidos de RNA crean un aminoácido, y varios aminoácidos forman cadenas peptídicas que adquieren formas tridimensionales, como ser alfa hélices y/o hojas beta que terminan dando la estructura tridimensional final a la proteína. Es importante mencionar que cada proteína debe adquirir el ensamblaje estructural correcto para llevar a cabo su función biológica en la célula.
Desde que se descubrió que el ácido nucleico es el encargado de llevar la información genética de cada célula, nace el deseo científico de querer entender y descifrar el lenguaje que las células usan para comunicarse entre ellas.
En 1995, el virus de la influenza fue el primer genoma secuenciado usando el primer método de secuenciación de llamado Sanger. Este método usa nucleótidos marcados que cada vez que se unen a sus respectivas contrapartes (A-T y/o G-C) emiten una señal de luz que es grabada por el equipo. El método de Sanger es considerado como la primera tecnología de secuenciación y fue utilizado para elucidar el primer genoma humano publicado el 2003, sin embargo, la desventaja de este método es que toma mucho tiempo para llevar a cabo la secuenciación de fragmentos largos de ácido nucleico y además tiene un costo elevado; de hecho, la secuencia del primer genoma humano llegó a costar 3 billones de dólares.
Ventajosamente el siglo XXI es considerado como el siglo de la revolución genómica, ya que se logran desarrollar nuevas tecnologías de secuenciación que permite secuenciar rápidamente genomas completos de una variedad muy grande de especies y a bajo costo, llegando a un precio de secuenciación de 100 dólares por genoma.
En la Figura 2 se describe el proceso de secuenciación genómica de cualquier organismo que posea ácido nucleico como parte de su estructura molecular, desde un virus (organismo no considerado vivo), pasando por las bacterias que son microorganismos procariotas (que no poseen un núcleo definido), hasta llegar a las células animales que son organismos eucariotas más complejos (organismos con núcleo definido). La secuenciación de la información genómica empieza con la extracción del ácido nucleico que será procesado según la tecnología de secuenciación que se elija. Una de estas tecnologías de segunda generación es la desarrollada por la empresa norteamericana Illumina, que mejoró la base científica usada en la secuenciación de Sanger, volviéndola más rápida y proporcionando la posibilidad de secuenciar muchos genomas al mismo tiempo de una forma muy sensitiva (sin cometer error) y con una gran cobertura. La única desventaja de la tecnología de Illumina es que el genoma antes de ser secuenciado debe ser cortado en pedazos cortos lo que obliga a tener un genoma de referencia para poder ensamblar las partes secuenciadas, lo cual es algo similar como armar un rompecabezas con piezas pequeñas donde es de mucha ayuda tener una foto del rompecabezas completo.
Otra tecnología alternativa o complementaria a la de Illumina es la tecnología de tercera generación que viene de la empresa inglesa Oxford Nanopore, empresa que usó nanotecnología para diseñar nano-poros que dejan pasar pedazos largos de ácido nucleico y la señal de detección de cada nucleótido se la realiza mediante corriente eléctrica, lo cual la vuelve en una secuenciación rápida en tiempo real. Y como si esto no fuera poco, la belleza de los equipos diseñados por Oxford Nanopore son de carácter portátil, es así como la NASA evalúo al MinION, la máquina de secuenciación más pequeña de esta empresa, en la Estación Espacial Internacional probando que es de fácil uso y muy eficiente llevando a cabo la secuenciación en el espacio exterior. El costo de esta tecnología tampoco es un problema ya que se pueden encontrar modelos desde unos mil dólares.
Y por último cabe mencionar que durante estos últimos años también existió un desarrollo de nuevos programas bioinformáticos para el análisis de las secuencias genómicas, algo que va de la mano con el avance de la tecnología de secuenciación. Estos programas bioinformáticos son tremendamente importantes en la visualización de la información genómica por que así logramos entender mejor la decodificación y/o elucidación del lenguaje que el ácido nucleico posee. Al visualizar los resultados de la secuenciación genómica se puede evaluar, por ejemplo, qué tan grande es el genoma del organismo secuenciado (bacteria, planta, virus, entre otros), qué genes se encuentran presentes en el ácido nucleico y qué proteínas codifican estos genes y las funciones que éstas cumplen en la célula. También se puede estudiar relaciones simbióticas entre comunidades microbianas asociadas a plantas o a los seres humanos, así se puede medir el impacto que pueden llegar a tener esclareciendo la organización estructural del microbioma y su nivel de biodiversidad, esto se logra al comparar la abundancia de las bacterias y la similitud entre sus genomas (1). En la actualidad también se usa un método llamado transcriptoma, que es una forma muy práctica para identificar genes que se activan en las células en cierto estado metabólico de la célula. En este método primero se extrae el RNA de las células y se los convierte a ADN para su secuenciación, es así como se logran identificar nuevas rutas metabólicas y/o genes que podrían ser claves en la cura de enfermedades.
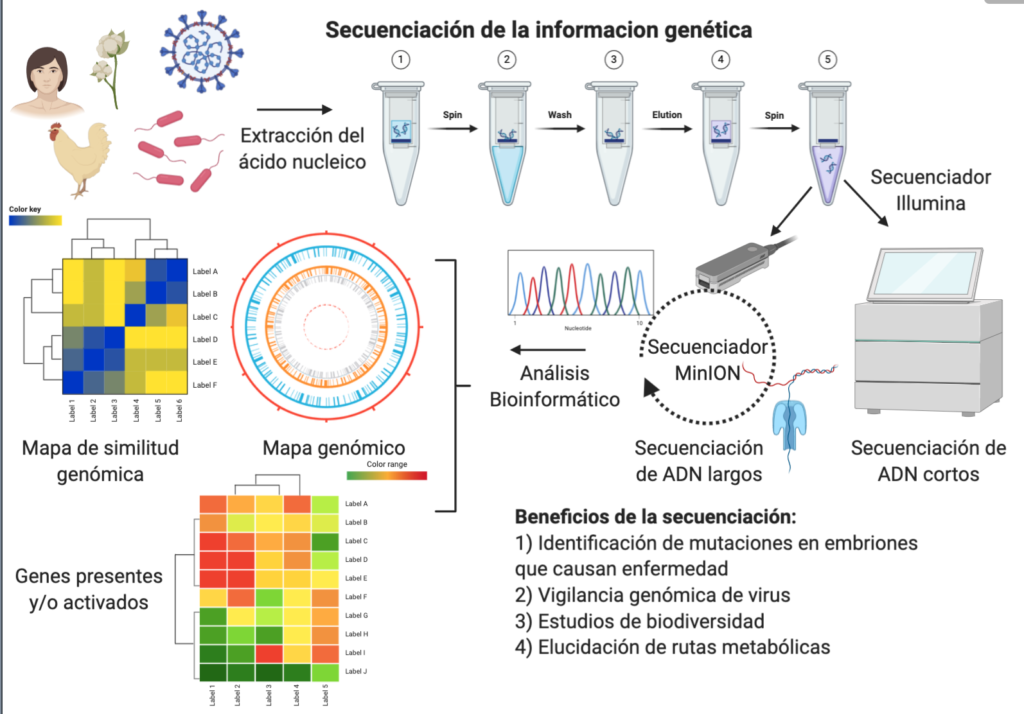
En la secuenciación de la información genómica, se empieza extrayendo el material genómico de la célula de interés, luego se prepara la librería genómica según la tecnología que se desea utilizar, por ejemplo, puede ser Illumina o Oxford Nanopore, una vez secuenciada la información genética viene la parte del análisis de los datos usando programas de bioinformática.
Este rápido avance en las tecnologías de secuenciación genómica, también conocidas como NGS sigla que viene de las palabras en inglés Next Generation Sequencing, abrieron la puerta hacia el área de la salud personalizada como el futuro de la medicina, ya que la NGS ofrece la posibilidad de secuenciar partes o todo el genoma y así identificar genes dañados o mutados en forma sensitiva (exacta) y rápida, haciendo que el diagnóstico y el monitoreo del tratamiento de enfermedades sean más efectivos (2).
En esta época de la pandemia por el SARS-CoV-2 se usa la NGS para monitorear cómo el virus va mutando de continente en continente o de persona en persona, esto se conoce como vigilancia genómica, y usándolo se reveló por ejemplo que en el norte de California existían varios tipos de virus que fueron introducidos por personas que venían de diferentes continentes (3). EL NGS también puede ser usado como una herramienta no invasiva en la selección de embriones saludables cuando los padres usan fertilización in vitro (4). Muchas veces los embriones se ven saludables en el microscopio, pero podrían ser portadores de mutaciones en genes que se expresan como síndromes, malformaciones o deficiencias autoinmunes en el niño, para evitar esto es recomendable secuenciar los embriones candidatos y elegir al más saludable para ser implantado en la fertilización in vitro (2).
Los países desarrollados como ser Estados Unidos, países europeos y China, ahora empezaron a invertir mucho dinero en empresas que ofrecen el servicio de secuenciación genómica y a la par están estableciendo leyes y regulaciones para que la normativa se encuentre clara en cada país. Estos años venideros también experimentaremos una explosión de bioemprendimientos que están surgiendo en diferentes países y que tienen el objetivo de explotar la información genética encontrada en la naturaleza. Ya existen muchas empresas apostando al potencial de la secuenciación de la información genómica y que tendrán aplicación en toda área imaginable, y podemos citar a la bio-foundry de ginkgo bioworks como un buen ejemplo de bioemprendimiento que recibió mucho dinero de inversores como ser Bayer empresa alemana en biotecnología. Los países en desarrollo, también tendrán la oportunidad de acceder a las tecnologías de NGS, esto debido a sus bajos costos actuales, que abren la posibilidad a la democratización de la tecnología a todo nivel de la sociedad. En el caso de Bolivia buscamos la mejor estrategia para transferir la tecnología de NGS a la juventud boliviana que está sedienta del conocimiento científico, es por eso que creamos iGEM Bolivia, una organización sin fines de lucro donde empezamos a transferir conocimiento a bolivianos en el área de biotecnología y biología sintética. Así, tenemos planeado trabajar en coordinación con universidades locales en cuatro departamentos y con mentores bolivianos científicos que viven en el extranjero (5). Identificamos que esta es la mejor estrategia para sobrellevar las limitaciones científicas que se viven en el país y pensamos mejorar la investigación científica a todo nivel, para eso tenemos los siguientes objetivos: ayudar a las universidades locales a establecer mejores laboratorios prácticos en biotecnología, enseñar ciencia básica a la sociedad boliviana y brindar cursos en bioética y bioseguridad a instituciones gubernamentales.
Juntos trabajaremos en grupos multidisciplinarios a nivel nacional para que empiecen a nacer los primeros bioemprendimientos bolivianos en el área de biorefinerías, aprovechando los recursos naturales y la biodiversidad microbiana que existe en Bolivia. Es aquí donde también necesitamos motivar a los empresarios para que empiecen a invertir en bioemprendimientos bolivianos que usen nuevas tecnologías y que lleven productos al mercado en base a la investigación y desarrollo. Finalmente, es muy importante remarcar que el progreso de la ciencia en países en desarrollo no solo dependerá de las ganas de aprender de los jóvenes bolivianos sino que también se necesitará un apoyo enorme por parte del gobierno y es realmente muy importante que los líderes políticos de turno entiendan que sin ciencia no existe futuro para Bolivia, puesto que si no hay un apoyo a la transferencia de tecnología mediante las políticas estatales, la sociedad boliviana seguirá rezagada y no tendrá acceso al uso de las nuevas tecnologías que seguirán avanzando a pasos agigantados en todo el mundo.
Referencias:
- https://www.biorxiv.org/content/10.1101/2020.04.27.064337v1.full
- https://www.cell.com/action/showPdf?pii=S0092-8674%2813%2901141-0
- https://science.sciencemag.org/content/early/2020/06/05/science.abb9263
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5081593/
- Contacto para unirse a iGEM Bolivia: tania.pozzo@gmail.com
